por Jason Z Zhang
Jason Zhang Es becaria postdoctoral Helen Hay Whitney HHMI en el laboratorio de David Baker en la Universidad de Washington. Su investigación se centra en la IA. de novo Diseño de proteínas para comprender la transducción de señales.
Medir los niveles de Ras activo en células vivas plantea un desafío formidable debido a su baja abundancia en las células y la naturaleza dinámica de la señalización de Ras. Ras sufre un rápido ciclo entre estados activos (ligados al GTP) e inactivos (ligados al PIB), lo que exige técnicas que capturen estas rápidas transiciones. Los ensayos bioquímicos tradicionales a menudo implican lisis celular y carecen de la capacidad de realizarse «en bioquímica celular», donde los matices espaciales y temporales de Ras se capturan dentro de células intactas. Además, la gran diversidad de isoformas de Ras complica aún más las mediciones, ya que cada isoforma exhibe distintos mecanismos reguladores. Las técnicas de imagen avanzadas, como la transferencia de energía por resonancia de fluorescencia (FRET) o la transferencia de energía por resonancia de bioluminiscencia (BRET), ofrecen información sobre la dinámica de Ras dentro de las células vivas.1,2. Sin embargo, estos métodos no están exentos de limitaciones, ya que pueden interferir con los procesos celulares normales o carecer de la sensibilidad necesaria para detectar fluctuaciones sutiles, ya que normalmente requieren una sobreexpresión de Ras para ver cambios significativos en la lectura. Por lo tanto, la dificultad para medir la actividad de Ras en células vivas surge de la necesidad de equilibrar la sensibilidad, la precisión temporal y la resolución espacial, lo que empuja a los investigadores a innovar continuamente en la búsqueda de una comprensión integral de esta vía de señalización basic.
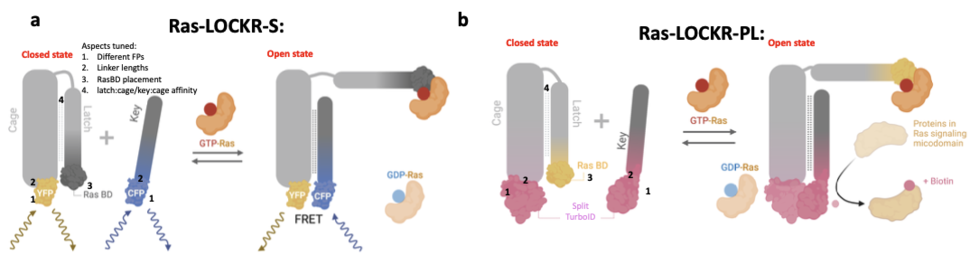
En nuestro trabajo reciente3mostramos que el interruptor de proteína LOCKR diseñado de novo4–6y las metodologías de diseño de proteínas computacionales basadas en IA detectan de manera confiable la activación de Ras dentro del rango fisiológicamente relevante de niveles endógenos de Ras-GTP, que ha eludido esfuerzos previos de diseño de biosensores. Se utilizaron proteínas de novo (proteínas que no tienen secuencia ni similitud estructural con las proteínas nativas) como nuevos soportes sensores, ya que podemos mutar estos dominios proteicos sin sacrificar la especificidad o funcionalidad porque son ortogonales al sistema biológico de interés. Los métodos de diseño de proteínas y las predicciones estructurales (AlphaFold) de estos interruptores de novo in silico predijeron qué mutaciones en las proteínas de novo alterarían la sensibilidad del sensor, que luego se probaron experimentalmente. Así, estos métodos computacionales guiaron el diseño y optimización del sensor. Con esta metodología de diseño, creamos dos sensores. (Figura 1):
1. Ras-LOCKR-S basado en fluorescencia que mide la actividad Ras (niveles de Ras-GTP) con las siguientes propiedades:
a. Medición en tiempo actual
b. Puede usarse en células vivas.
C. Resolución unicelular
d. Dirigido a diferentes regiones subcelulares para medir la actividad Ras en compartimentos subcelulares particulares
2. Etiquetador de proximidad dependiente de la actividad Ras Ras-LOCKR-PL que perfila el entorno alrededor de Ras-GTP con las siguientes propiedades:
a. Perfila entornos Ras activos en 4 horas
b. Puede usarse en resolución unicelular si se combina con ensayos de ligadura de proximidad7
C. Dirigido a diferentes regiones subcelulares para medir la actividad Ras en compartimentos subcelulares particulares
Utilizando estos nuevos sensores, obtuvimos información sobre los mecanismos de señalización oncogénica y descubrimos que el Ras endógeno está activo no solo en el PM sino también en las endomembranas (Golgi) y en los gránulos citosólicos sin membrana cuando se fusiona una oncoproteína tirosina quinasa receptora ( EML4-Alk) se expresó. Dirigiendo nuestras herramientas Ras-LOCKR a gránulos que contienen EML4-Alk8 (observado en adenocarcinomas de pulmón) permitió el descubrimiento de varios factores imprevistos dentro de estos gránulos. Identificamos a SAM68 como un efector ascendente de la actividad citosólica aberrante de Ras dentro de los gránulos de EML4-Alk, proporcionando así una imagen más completa de cómo se puede activar Ras y es capaz de impulsar la señalización oncogénica en ausencia de membranas. Nuestro hallazgo de que la co-inhibición de SAM68 y Alk conduce a una mayor muerte de las células cancerosas y a la inhibición de la actividad Ras medida con Ras-LOCKR-S sugiere que el tratamiento conjunto con inhibidores de Alk y SAM68 podría ayudar a superar la resistencia a los medicamentos en el pulmón impulsado por EML4-Alk. cáncer.
En basic, estos resultados ilustran el poder de nuestra metodología de desarrollo de sensores para generar herramientas que mapeen las actividades, mecanismos y funciones de moléculas fisiológicamente relevantes como Ras. Estos sensores Ras finalmente están permitiendo a los investigadores interrogar la dinámica de la actividad Ras en su contexto celular nativo. Imaginamos que las herramientas descritas aquí se pueden utilizar para responder muchas preguntas urgentes en los campos del cáncer y la señalización de Ras.
Nota: los plásmidos que codifican Ras-LOCKR-S y Ras-LOCKR-PL están disponibles en Addgene: https://www.addgene.org/browse/article/28244240.