En este estudio, incluimos a 1351 personas sin infección previa por SARS-CoV-2, que recibieron las dos dosis de la vacuna BNT162b2 COVID-19 y fueron reclutadas en tres hospitales italianos: Fondazione IRCCS Istituto Neurologico Carlo Besta en Milán (norte= 306), Azienda Ospedaliero-Universitaria Senese en Siena (norte= 689), y Fondazione IRCCS Casa Sollievo della Sofferenza, San Giovanni Rotondo (FG) (norte= 356; Mesa 1). El período de reclutamiento abarcó desde el 27 de diciembre de 2020 y el 15 de mayo de 2021. Los participantes de origen europeo, según lo determinado por el análisis de componentes principales (PCA, figura complementaria. 1) fueron incluidos en los análisis genéticos. La cohorte estaba formada por vacunados italianos que eran principalmente trabajadores hospitalarios, con una representación femenina predominante (66,5%) y una edad media de 48 años (rango: 19-84). La medición de los niveles de IgG se realizó en una mediana de tiempo de 40 días después de la administración de la segunda dosis de vacuna (rango intercuartílico, RIC = 68). Los niveles de IgG oscilaron entre 12,64 y 6.056 BAU/ml, con una mediana de 801,5 BAU/ml.
Las tres collection fueron bastante diferentes en términos de distribución por sexo (prueba de chi cuadrado PAG-valor = 0,0004): de hecho, aunque el sexo femenino fue predominante en las tres collection, Fondazione IRCCS Casa Sollievo della Sofferenza, San Giovanni Rotondo (en adelante SGR) reclutó más sujetos masculinos (~42%) que los otros centros de reclutamiento (aproximadamente 30%). Las tres collection también diferían en cuanto a la distribución por edades (prueba de Kruskal-Wallis PAG-valor < 0,0001) siendo los individuos SGR mayores que los demás y la serie de Siena teniendo el valor de edad mediana más bajo. La diferencia más relevante entre las collection se refiere al intervalo de tiempo (en días) entre la segunda dosis de vacunación y la medición serológica de IgG (check de Kruskal-Wallis). PAG-valor < 0,0001). De hecho, los niveles de IgG en la serie de Siena no se midieron 30 ± 5 días después del refuerzo de la vacuna, como sí se hizo en las otras dos collection.
Dado que los valores de IgG no se distribuyeron normalmente (prueba de Shapiro-Wilk PAG -valor < 0,0001), aplicamos la transformación regular inversa a estos valores (Fig. 3). Luego realizamos un modelo de regresión lineal multivariable entre valores normalizados de IgG, edad, sexo, centros de reclutamiento y tiempo entre la vacunación y la medición de los niveles de IgG (Tabla 2). Los valores de IgG normalizados se correlacionaron inversamente con la edad en el momento de la vacunación y el tiempo (en días) transcurrido entre la vacunación y la recolección de suero para la medición de anticuerpos. La cantidad de anticuerpos, en cambio, no fue significativamente diferente entre mujeres y hombres. Como period de esperar, observamos niveles más bajos de IgG en individuos de Siena que los reclutados en las otras dos cohortes; los niveles medios de IgG de los sujetos de Siena diferían en 902,5 y 1.078,5 BAU/ml de la mediana de los individuos de Milán y SGR, respectivamente. Sin embargo, es importante señalar que, cuando consideramos solo aquellos sujetos cuyos anticuerpos se midieron 30 ± 5 días después de la segunda dosis de la vacuna, no observamos diferencias significativas en los niveles de IgG entre los tres grupos de individuos de los tres centros (valores medios : 1160, 1395 y 1489 en Siena, Milán y SGR, respectivamente; prueba de Kruskal-Wallis PAG-valor > 0,05). Esto sugiere que las diferencias entre los tres centros se debieron más probablemente a las discrepancias en el tiempo entre la vacunación y la medición de IgG, que a diferencias entre operadores y pruebas.
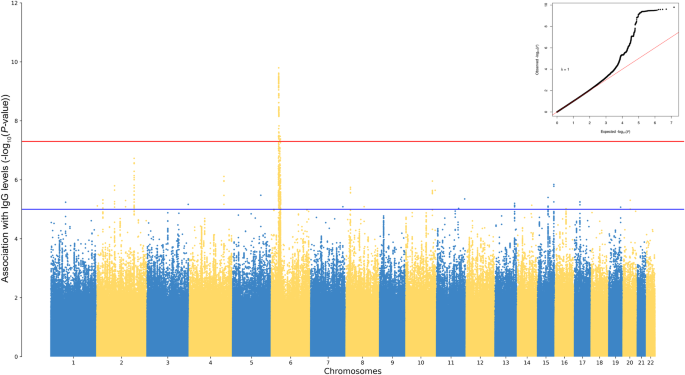
Llevamos a cabo un análisis de asociación de todo el genoma entre los niveles de IgG normalizados y los genotipos imputados de 7.339.393 variantes, incluido en el modelo de regresión lineal el sexo, la edad en el momento de la vacunación, el centro de reclutamiento, los primeros 5 componentes principales (PC) y el intervalo de tiempo entre la segunda dosis de vacuna y la prueba serológica como posibles factores de confusión. Los resultados se presentan en el gráfico de Manhattan, que se muestra en la Fig. 1. Se identificó una señal estadísticamente significativa en el cromosoma 6, en el locus HLA, con 144 variantes asociadas a un valor nominal PAG -valor < 5,0 × 10−8 (Dato suplementario 1). Estas variantes abarcaban una región de 29,7 Mbp a 32,6 Mbp (según la estructura genómica humana GRCh37/hg19) y la variante principal, rs1632893, se mapeó menos de 4 kb por delante de la HLA-Agen (beta = 0,28, SE = 0,044, IC del 95%: 0,20 – 0,37, PAG-valor = 1,6 × 10−10). También se observó una señal sugestiva por debajo del umbral de significación de todo el genoma en el cromosoma 2. La variante principal, rs11692649 en la posición 183.266.641, es una variante intrónica de PDE1Agen (beta = −0,17, SE = 0,033, IC del 95%: −0,24 a −0,11, PAG-valor = 1,87 × 10−7).
Gráfico de Manhattan de los resultados del GWAS entre los valores de IgG anti-pico normalizados inversamente y 7.339.393 variantes imputadas, probados en un modelo de regresión lineal, utilizando sexo, edad en el momento de la vacunación, centro, los primeros 5 componentes principales (PC) y el tiempo. intervalo entre la segunda dosis de vacuna y la prueba serológica como covariables. Los SNP se trazan en el eje x según su posición genómica (GChr37, liberación de hg19), y PAG -valores (−log10PAG) por su asociación con los niveles de IgG en el eje y. La línea roja horizontal representa el umbral de importancia para todo el genoma (PAG -valor < 5,0 × 10−8). En la esquina superior derecha se muestra el gráfico QQ de las variables observadas y esperadas. PAGvalores. Se informa el issue de inflación genómica (λ).
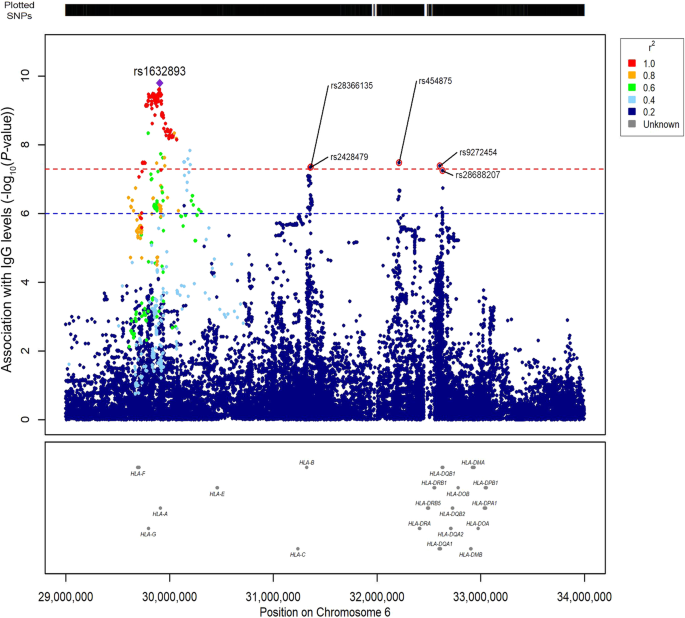
Al acercarnos al locus del cromosoma 6, observamos que no había una sola señal de asociación (Fig. 2). De hecho, hubo otras regiones, además de la liderada por rs1632893 en la posición 29.905.193, aunque este locus tiene el mayor número de variantes significativamente asociadas (norte= 140), abarcando desde 29,7 Mbp hasta 30,2 Mbp. Para todas estas variantes, que están en alto (r2> 0,7) desequilibrio de ligamiento entre sí (excepto ocho de ellos), el aumento del número de alelos menores en los genotipos se asoció con niveles más altos de IgG. Un pico adicional de asociación (beta = 0,27, SE = 0,048, IC del 95%: 0,17 – 0,36, PAG-valor = 4,5 × 10−8) fue liderado por rs28366135 (en la posición 31,364,105), que asigna menos de 40 kbp en sentido ascendente HLA-Bgen, junto con rs2428479 en la posición 31.361.110 (beta = 0,27, SE = 0,048, IC del 95 %: 0,17 – 0,36, PAG-valor = 4,6 x 10−8). También para estas variantes, observamos que los individuos homocigotos para los alelos menores o heterocigotos tenían niveles más altos de IgG que los homocigotos para los alelos mayores. Luego, observamos otras dos variantes (rs454875, beta = −0,23, SE = 0,042, IC del 95%: −0,32 a −0,15, PAG -valor = 3,4 × 10−8; rs9272454, beta = −0,17, SE = 0,031, IC del 95 %: −0,23 a −0,11, PAG-valor = 4.2×10−8) en las posiciones 32.213.008 y 32.605.525, respectivamente. Este último period una variante intrónica de HLA-DQA1. Otra variante cercana a estas dos y justo por debajo del umbral de significación de todo el genoma fue rs28688207 (beta = −0,28, SE = 0,052, IC del 95 %: −0,38 a −0,18, PAG-valor = 5,5 × 10−8), una variante del aceptador de empalme de HLA-DQB1gene. A diferencia de las variantes anteriores, los alelos menores de estas tres últimas se asociaron significativamente con los niveles más bajos de IgG.
Gráfico ampliado del locus en el cromosoma 6 identificado en el GWAS. Las parcelas abarcan la región de 29 Mbp a 33 Mbp y contienen genes HLA y todas las variantes imputadas analizadas. Los SNP se trazan en el eje x según su posición en el cromosoma 6, y PAG -valores (−log10PAG) para su asociación con los niveles de IgG se representan en el eje y. La línea discontinua roja horizontal representa el umbral de importancia (PAG -valor < 5,0 × 10−8) mientras que el azul representa un umbral sugerente (PAG-valor < 1,0 × 10−6 ). El colour del punto representa el nivel de desequilibrio de ligamiento, expresado como r2 entre cada SNP y la variante principal (rs1632893, diamante morado).
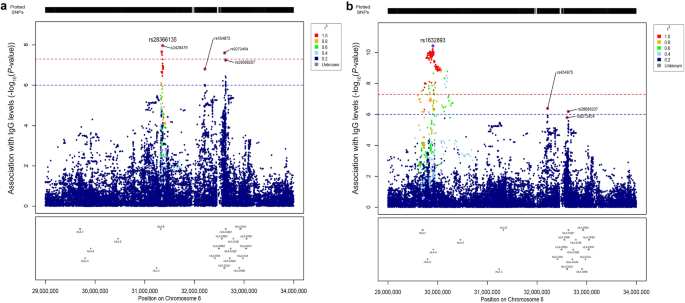
Calculamos el desequilibrio de ligamiento (LD) entre rs1632893 y las variantes de las otras dos regiones, y observamos que no estaban en LD (D’ = 0,14 y r2 = 0,00033 con rs28366135 y rs2428479; D’ = 0,34 y r2= 0,0026 con rs454875, D’ = 0,0020 y r2= 1,1 × 10−6con rs9272454, y D’ = 0,086 y r2 = 9,4×10−5 con rs28688207; Dato suplementario 1), lo que sugiere que las tres señales son independientes. De hecho, en un análisis de regresión lineal que prueba el mismo modelo anterior, pero con el genotipo de rs1632893 como covariable adicional, observamos que las otras señales (rs28366135, rs2428479, así como otras variantes en HLA-B locus y rs9272454 en HLA-DQA1 ) siguió siendo estadísticamente significativo (Fig. 3A). En cambio, se realizó el mismo análisis utilizando rs28366135 como covariable: en este caso solo las señales de asociación en el HLA-Alocus, liderado por rs1632893 siguió siendo significativo (Fig. 3B). Finalmente, en un modelo de regresión lineal donde se agregaron los genotipos rs1632893 y rs28366135 a las otras covariables, la señal en el cromosoma 2 (rs11692649) se volvió estadísticamente significativa (beta = −0,18, SE = 0,032, IC del 95 %: −0,24 a −0,11, PAG-valor = 4,9 × 10−8), mientras que los SNP en HLA-DQA1y HLA-DQB1los genes tenían PAG-valores por debajo del umbral de significancia de GWAS (Fig. 4).
Los dos paneles muestran los resultados de los análisis ajustados para rs1632893 (a) o para rs28366135 (b). Las parcelas abarcan la región de 29 Mbp a 33 Mbp y contienen genes HLA y todas las variantes imputadas analizadas. Los SNP se trazan en el eje x según su posición en el cromosoma 6, y PAG-valores (−log10PAG) para su asociación con los niveles de IgG se representan en el eje y. La línea discontinua roja horizontal representa el umbral de importancia (PAG-valor < 5,0 × 10−8) mientras que el azul representa un umbral sugerente (PAG-valor < 1,0 × 10−6). El colour del punto representa el nivel de desequilibrio de ligamiento, expresado como r2entre cada SNP y la variante principal (diamantes morados).
Luego, imputamos alelos HLA de cuatro dígitos (norte= 204) utilizando los datos de genotipado de nuestros 1351 individuos e informamos sus frecuencias en nuestra serie en Datos complementarios 2. Analizamos, en un modelo de regresión lineal con las mismas covariables incluidas en el GWAS, la asociación entre los niveles de IgG normalizados y los alelos HLA. Observamos que 12 alelos HLA se asociaron significativamente con niveles de IgG en FDR <0,01 (Datos complementarios 2). Los dos alelos más significativos (es decir, HLA-A*03:01 y HLA-C*12:02) pertenecían al complejo mayor de histocompatibilidad (MHC) de clase I, junto con otros dos alelos (HLA-B*52:01 y HLA-A*29:02). En cambio, los otros ocho alelos significativamente asociados con los niveles de IgG fueron moléculas MHC de clase II (HLA-DQB1*06:01, HLA-DRB1*15:02, HLA-DQB1*02:01, HLA-DRB1*14:01, HLA -DQA1*01:01, HLA-DQA1*02:01, HLA-DQB1*05:03 y HLA-DRB1*07:01). Las frecuencias de estos alelos son bastante diferentes, como se esperaba (p. ej., HLA-DQB1*02:01, HLA-DQA1*01:01, HLA-DQA1*02:01, HLA-A*03:01 y HLA-DRB1 *07:01 fueron más frecuentes que HLA-C*12:02, HLA-DQB1*06:01, HLA-DRB1*15:02, HLA-DQB1*05:03, HLA-DRB1*14:01, HLA- B*52:01 y HLA-A*29:02). Dado que algunos de estos alelos estaban en LD, informamos el R2y valores D’, junto con sus frecuencias, en la tabla complementaria 1.